随着医学影像技术和计算机视觉技术的快速发展,医疗诊断中的自动化工具正逐渐成为临床应用中的研究热点。在肝脏肿瘤的早期检测与诊断中,传统的人工方法耗时较长,且容易受医生的主观经验影响,诊断结果的准确性和一致性难以保证。基于此,本研究结合深度学习技术,设计并实现了一个医学影像肝脏肿瘤病症检测与诊断系统,旨在提高诊断效率、减少误诊率,并为医生提供科学、直观的辅助工具。
本系统的核心采用了YOLOv8模型,一种当前性能较为领先的目标检测算法,用于识别医学影像中的肝脏及肿瘤区域。通过对大规模医学影像数据集进行标注与预处理,系统能够自动学习肝脏和肿瘤的特征,并精确定位图像中的病灶区域。相比传统的肿瘤检测方法,YOLOv8模型具有检测速度快、精度高的优势,能够在保持高召回率的同时大幅减少误报,满足实际临床应用中的实时性和准确性要求。
此外,系统集成了PyQt5框架开发的图形用户界面(GUI),该界面友好且易于操作,为医生提供了丰富的功能模块。医生可以通过界面方便地加载患者的医学影像,查看模型自动标注的肝脏和肿瘤区域,并获得系统生成的诊断报告。该系统还支持批量处理多张医学影像,极大提升了临床医生的工作效率。同时,界面提供了图像缩放、对比度调整等功能,方便医生在系统的基础上进行进一步的诊断分析,确保检测结果的可解释性和应用价值。
在模型的开发过程中,本文重点介绍了数据集的收集、预处理和标注方法,特别是如何通过数据增强、图像归一化等技术提高模型的泛化能力。此外,系统还结合了迁移学习和精细调参等深度学习优化策略,以确保模型在新数据上的表现优越。通过大规模的实验评估,结果表明,该系统在肝脏肿瘤检测任务上取得了卓越的性能,模型的平均精度(mAP)在多类目标检测中达到了极高的水平,且在肝脏及肿瘤区域的精确识别上具有稳定的表现。
总体而言,该基于深度学习的医学影像肝脏肿瘤检测与诊断系统在临床应用中具有广泛的潜力。它不仅能够提高医生的诊断效率,还能减少由于人为因素导致的误诊漏诊问题,为患者提供更准确、更及时的治疗方案
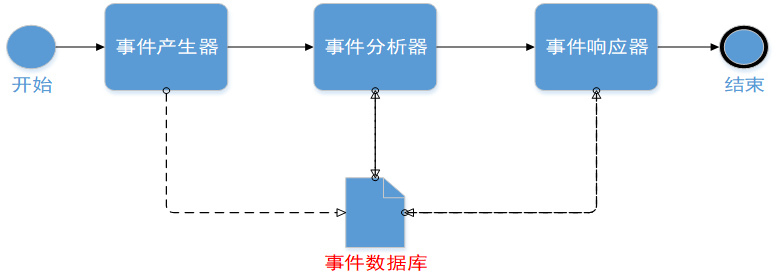
算法流程

项目数据
通过搜集关于数据集为各种各样的肝脏肿瘤病症相关图像,并使用Labelimg标注工具对每张图片进行标注,分3个检测类别,分别是’No Tumor’, ‘Tumor’, ‘Liver’。
目标检测标注工具
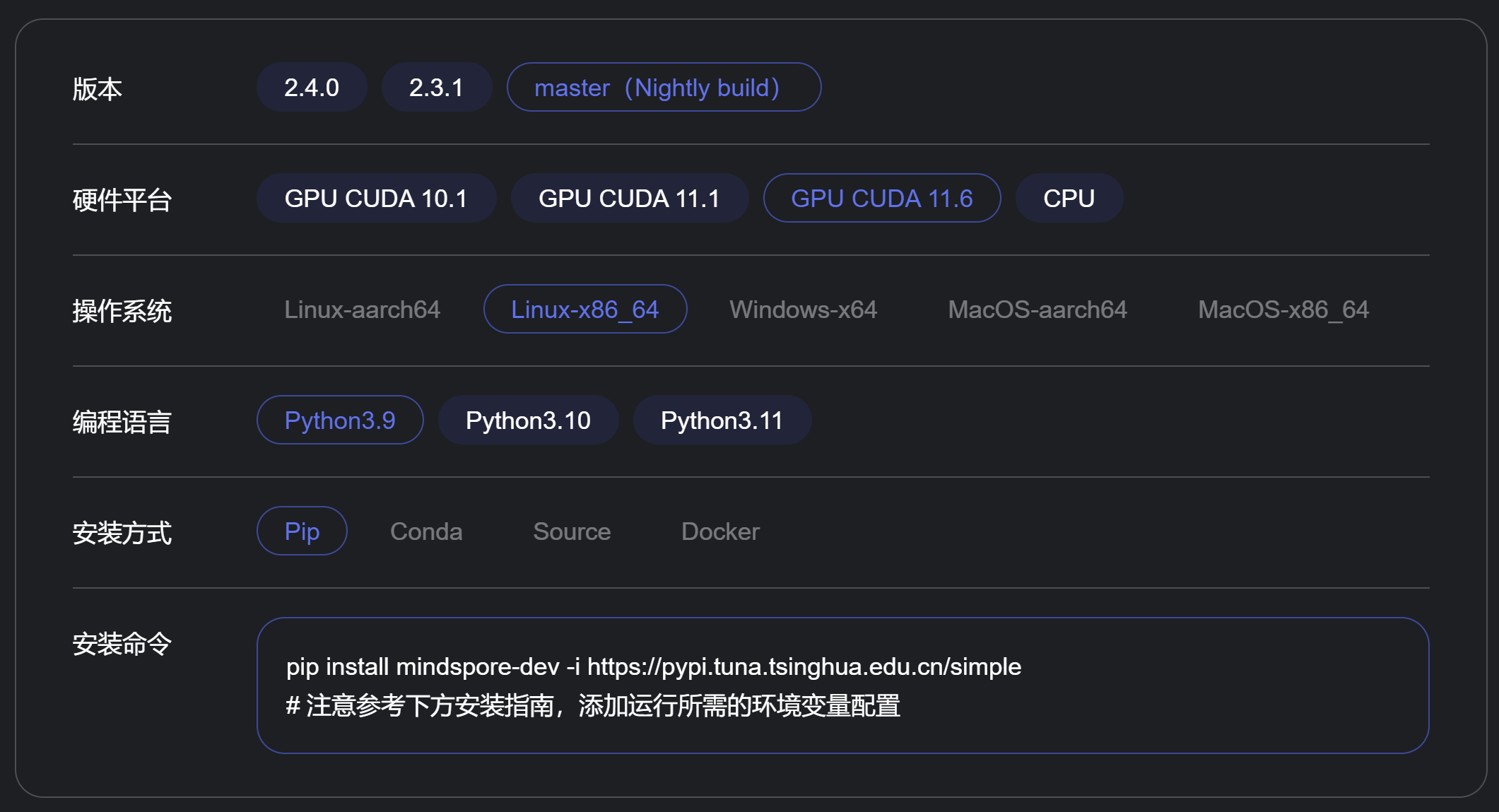
(1)labelimg:开源的图像标注工具,标签可用于分类和目标检测,它是用python写的,并使用Qt作为其图形界面,简单好用(虽然是英文版的)。其注释以 PASCAL VOC格式保存为XML文件,这是ImageNet使用的格式。此外,它还支持 COCO数据集格式。
(2)安装labelimg 在cmd输入以下命令 pip install labelimg -i https://pypi.tuna.tsinghua.edu.cn/simple
![]()
结束后,在cmd中输入labelimg
![]()
初识labelimg
打开后,我们自己设置一下
在View中勾选Auto Save mode
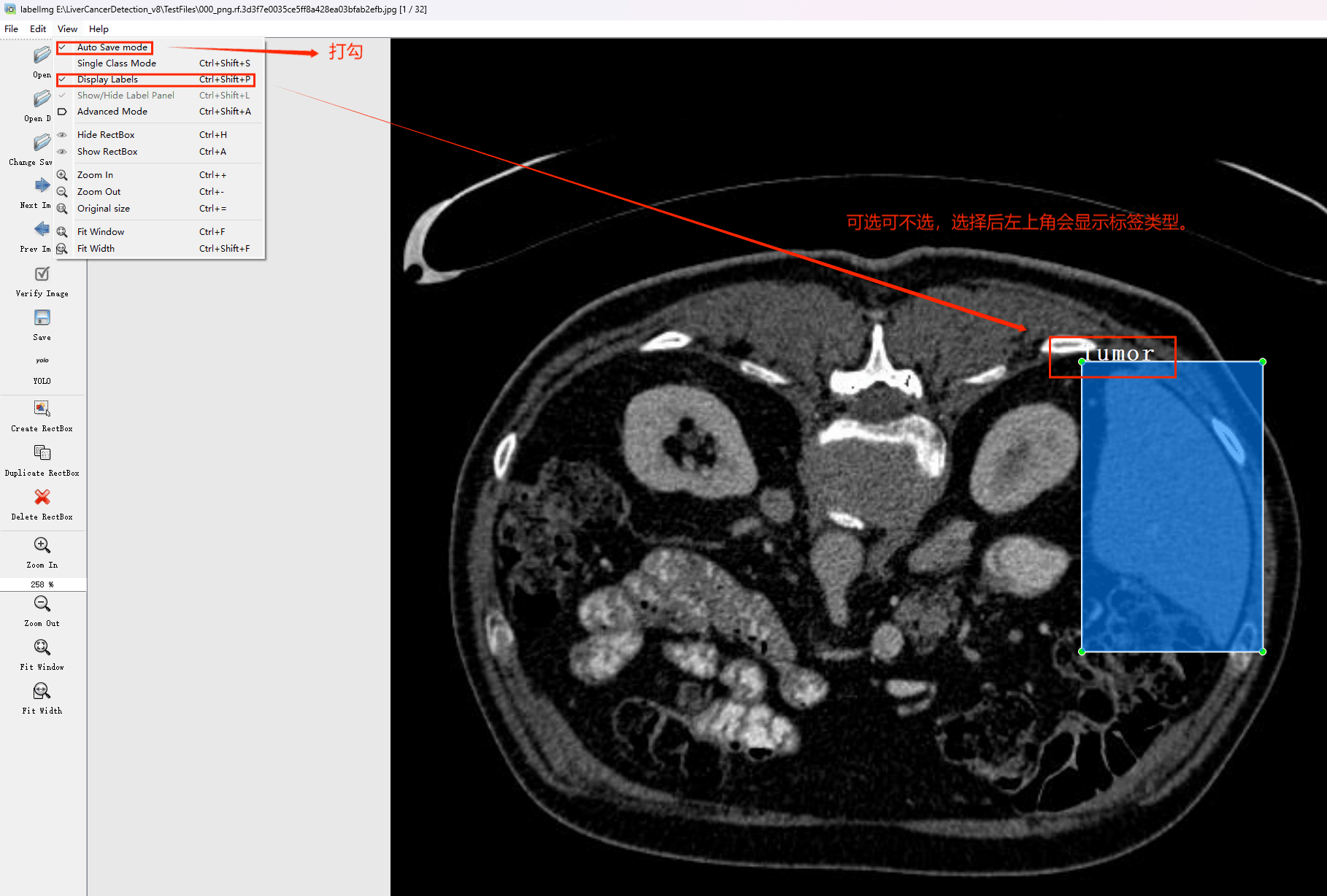
接下来我们打开需要标注的图片文件夹
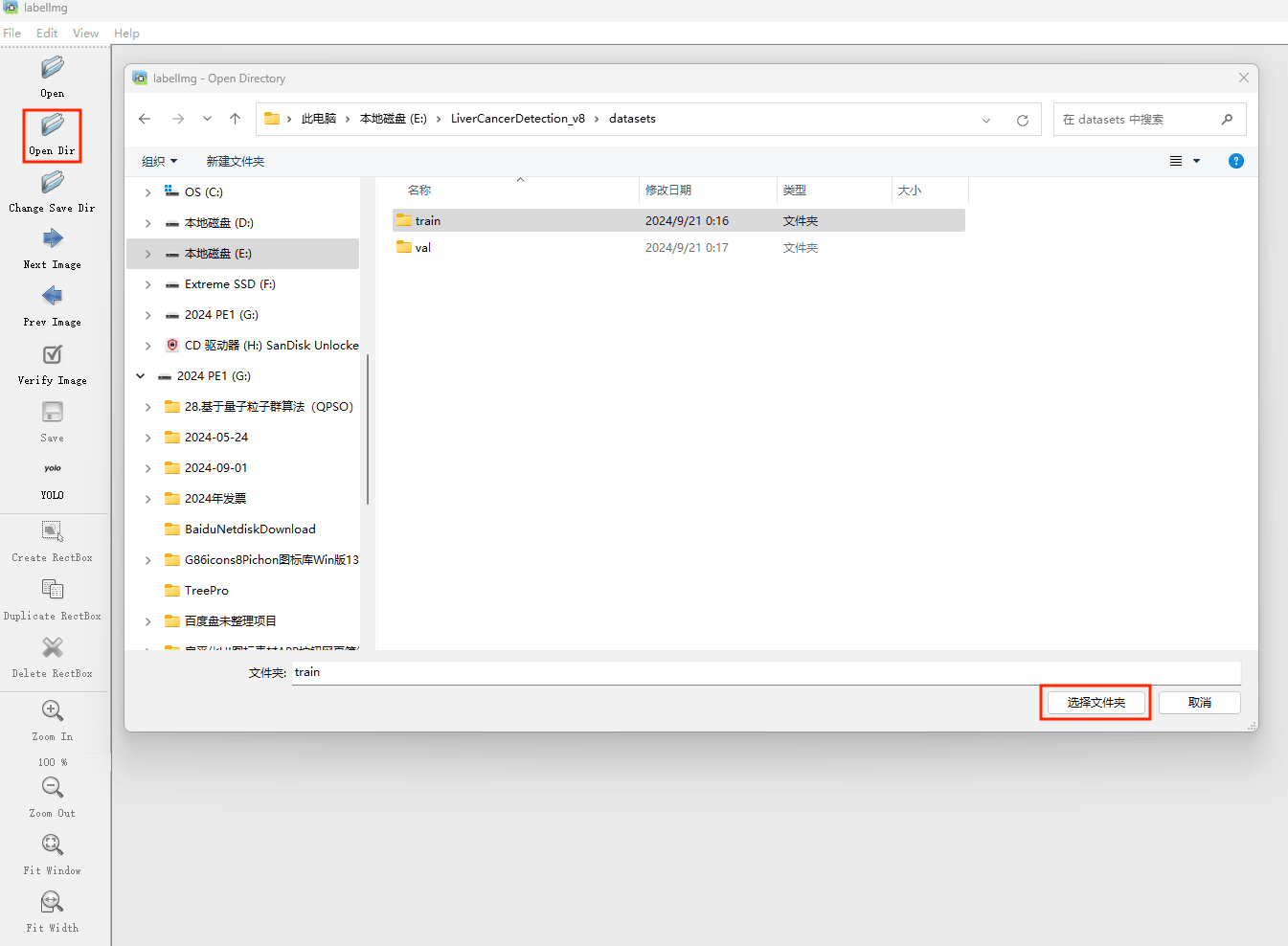
并设置标注文件保存的目录(上图中的Change Save Dir)
接下来就开始标注,画框,标记目标的label,然后d切换到下一张继续标注,不断重复重复。
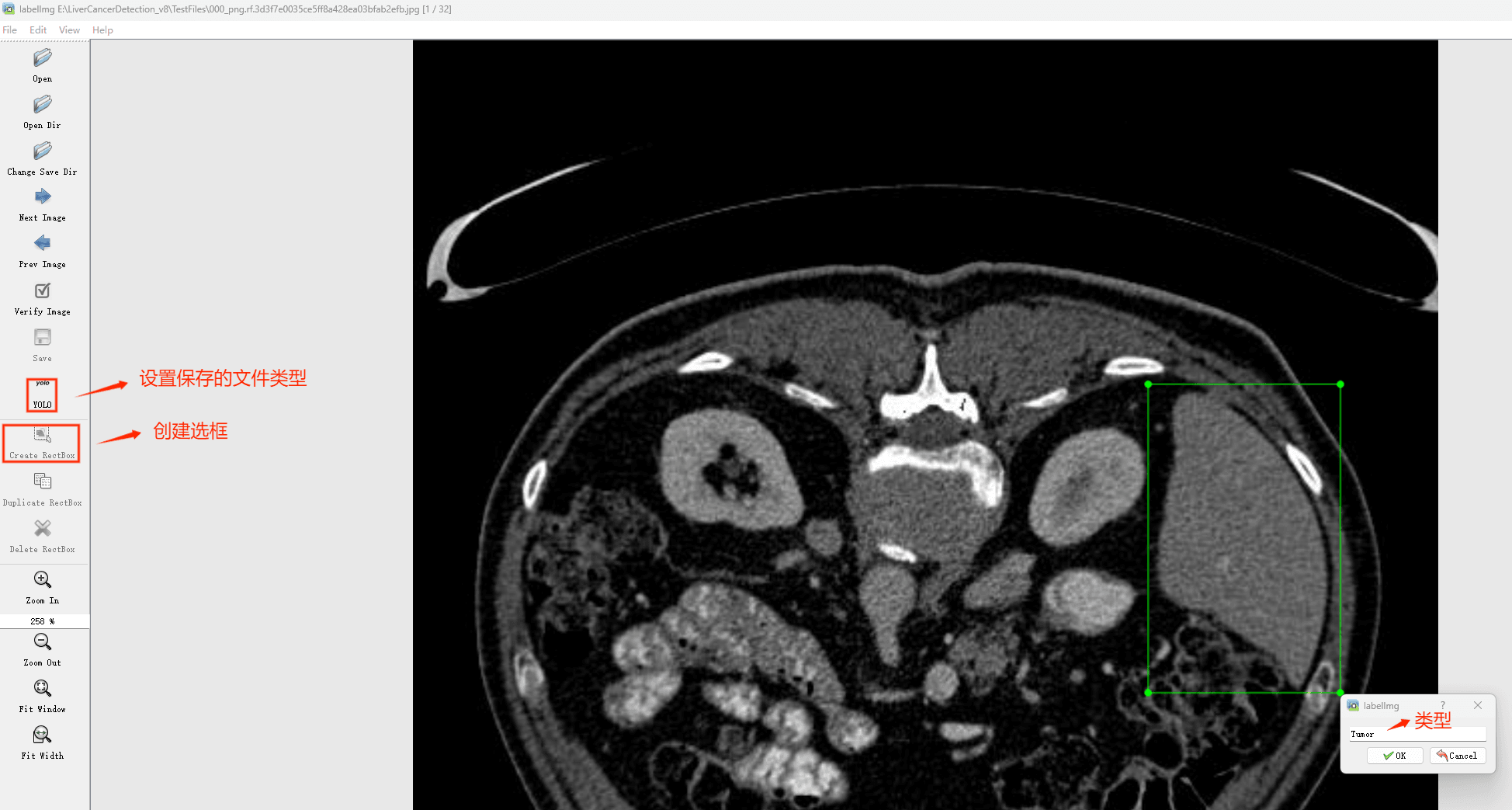
Labelimg的快捷键
(3)数据准备
这里建议新建一个名为data的文件夹(这个是约定俗成,不这么做也行),里面创建一个名为images的文件夹存放我们需要打标签的图片文件;再创建一个名为labels存放标注的标签文件;最后创建一个名为 classes.txt 的txt文件来存放所要标注的类别名称。
data的目录结构如下:
│─img_data
│─images 存放需要打标签的图片文件
│─labels 存放标注的标签文件
└ classes.txt 定义自己要标注的所有类别(这个文件可有可无,但是在我们定义类别比较多的时候,最好有这个创建一个这样的txt文件来存放类别)
首先在images这个文件夹放置待标注的图片。
生成文件如下:
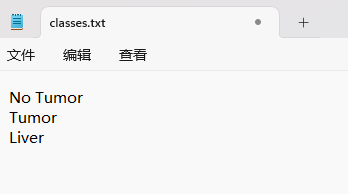
“classes.txt”定义了你的 YOLO 标签所引用的类名列表。
(4)YOLO模式创建标签的样式
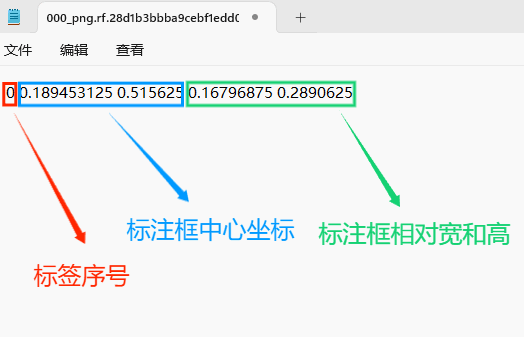
存放标签信息的文件的文件名为与图片名相同,内容由N行5列数据组成。
每一行代表标注的一个目标,通常包括五个数据,从左到右依次为:类别id、x_center、y_center、width、height。
其中:
–x类别id代表标注目标的类别;
–x_center和y_center代表标注框的相对中心坐标;
–xwidth和height代表标注框的相对宽和高。
注意:这里的中心点坐标、宽和高都是相对数据!!!
存放标签类别的文件的文件名为classes.txt (固定不变),用于存放创建的标签类别。
完成后可进行后续的yolo训练方面的操作。
模型训练
模型的训练、评估与推理
1.YOLOv8的基本原理
YOLOv8是一个SOTA模型,它建立在Yolo系列历史版本的基础上,并引入了新的功能和改进点,以进一步提升性能和灵活性,使其成为实现目标检测、图像分割、姿态估计等任务的最佳选择。其具体创新点包括一个新的骨干网络、一个新的Ancher-Free检测头和一个新的损失函数,可在CPU到GPU的多种硬件平台上运行。
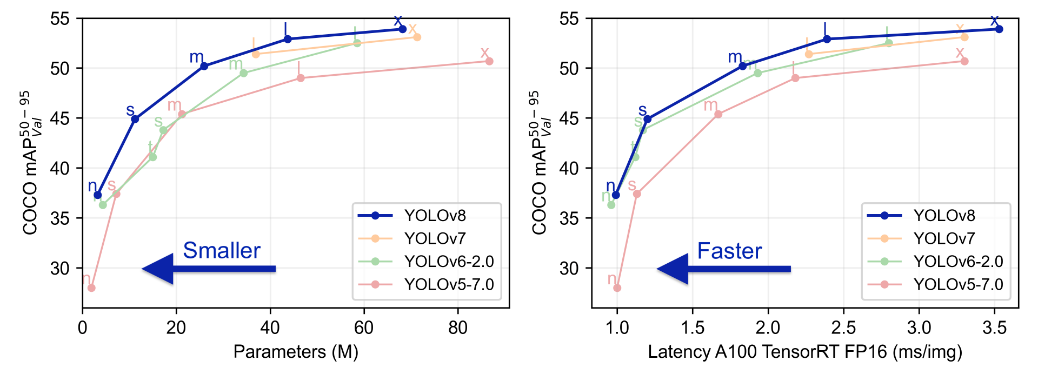
YOLOv8是Yolo系列模型的最新王者,各种指标全面超越现有对象检测与实例分割模型,借鉴了Yolov5、Yolov6、YoloX等模型的设计优点,在全面提升改进Yolov5模型结构的基础上实现,同时保持了Yolov5工程化简洁易用的优势。
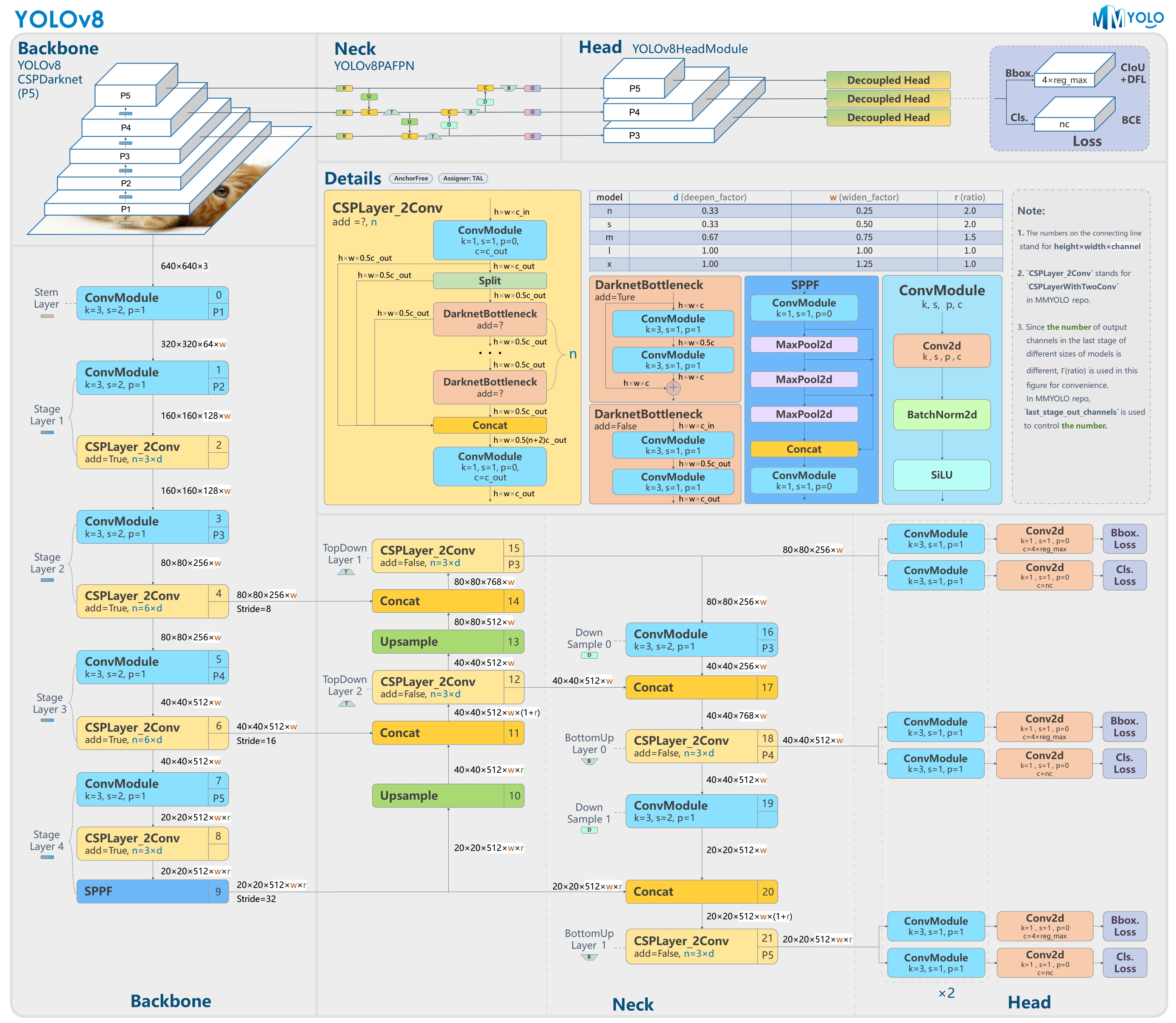
Yolov8模型网络结构图如下图所示:
2.数据集准备与训练
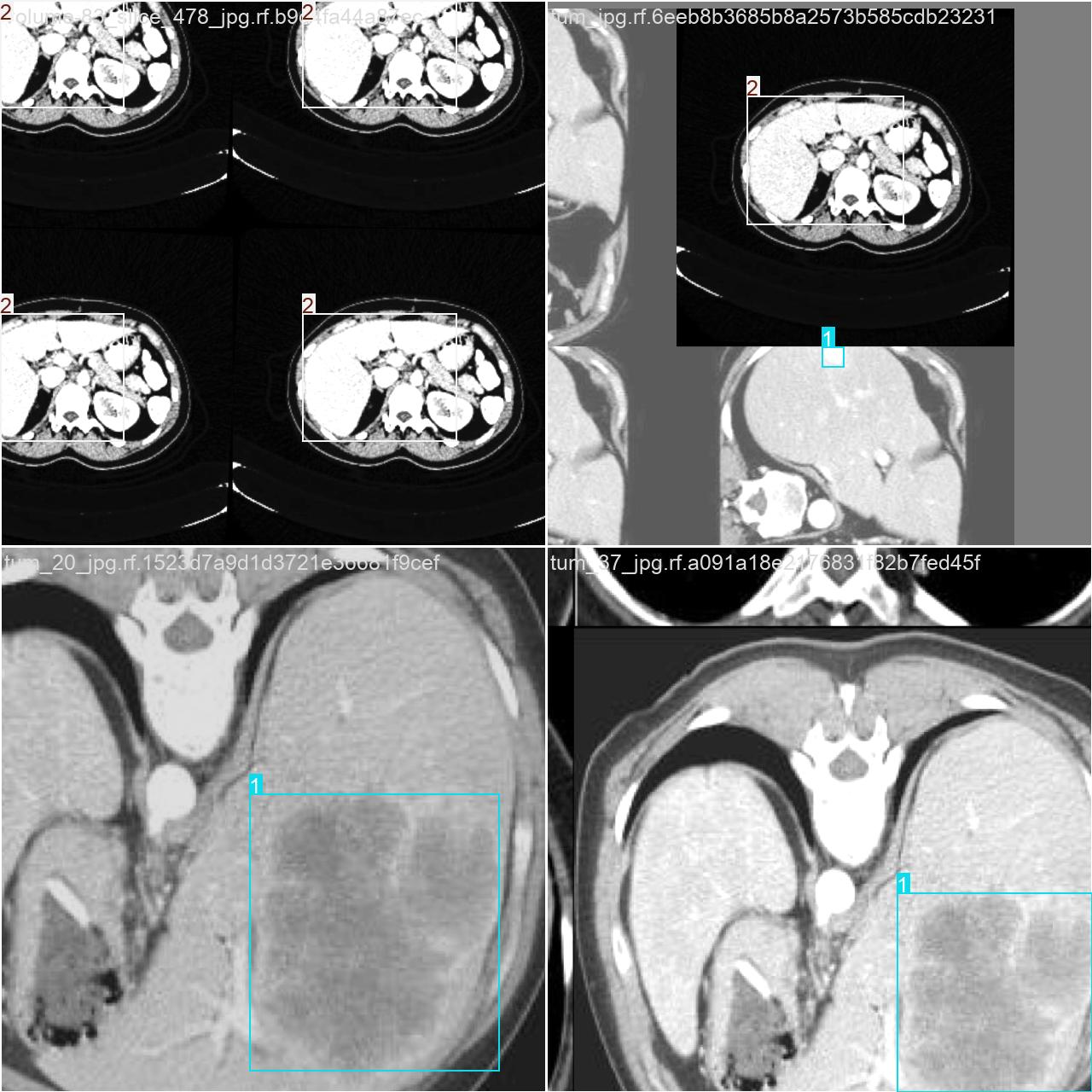
本研究使用了包含各肝脏病症相关图像的数据集,并通过Labelimg标注工具对每张图像中的目标边框(Bounding Box)及其类别进行标注。然后主要基于YOLOv8n这种模型进行模型的训练,训练完成后对模型在验证集上的表现进行全面的性能评估及对比分析。模型训练和评估流程基本一致,包括:数据集准备、模型训练、模型评估。本次标注的目标类别为肝脏病症,数据集中共计包含12800张图像,其中训练集占10171张,验证集占2629张。部分图像如下图所示:

部分标注如下图所示:
图片数据的存放格式如下,在项目目录中新建datasets目录,同时将检测的图片分为训练集与验证集放入datasets目录下。
接着需要新建一个data.yaml文件,用于存储训练数据的路径及模型需要进行检测的类别。YOLOv8在进行模型训练时,会读取该文件的信息,用于进行模型的训练与验证。
data.yaml的具体内容如下:
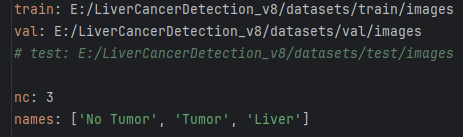
train: E:/LiverCancerDetection_v8/datasets/train/images 训练集的路径
val: E:/LiverCancerDetection_v8/datasets/val/images 验证集的路径
# test: E:/LiverCancerDetection_v8/datasets/test/images 测试集的路径
nc: 3 模型检测的类别数,共有个3类别。
names: [‘No Tumor’, ‘Tumor’, ‘Liver’]
这个文件定义了用于模型训练和验证的数据集路径,以及模型将要检测的目标类别。
数据准备完成后,通过调用train.py文件进行模型训练,epochs参数用于调整训练的轮数,batch参数用于调整训练的批次大小(根据内存大小调整,最小为1)。
CPU/GPU训练代码如下:
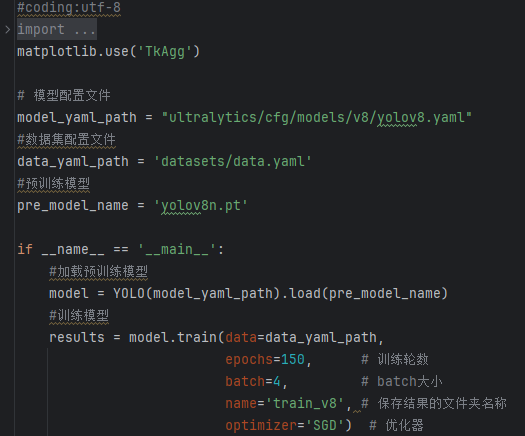
加载名为 yolov8n.pt 的预训练YOLOv8模型,yolov8n.pt是预先训练好的模型文件。
使用YOLO模型进行训练,主要参数说明如下:
(1)data=data_yaml_path: 指定了用于训练的数据集配置文件。
(2)epochs=150: 设定训练的轮数为150轮。
(3)batch=4: 指定了每个批次的样本数量为4。
(4)optimizer=’SGD’):SGD 优化器。
(7)name=’train_v8′: 指定了此次训练的命名标签,用于区分不同的训练实验。
3.训练结果评估
在深度学习的过程中,我们通常通过观察损失函数下降的曲线来了解模型的训练情况。对于YOLOv8模型的训练,主要涉及三类损失:定位损失(box_loss)、分类损失(cls_loss)以及动态特征损失(dfl_loss)。训练完成后,相关的训练过程和结果文件会保存在 runs/ 目录下,具体如下:
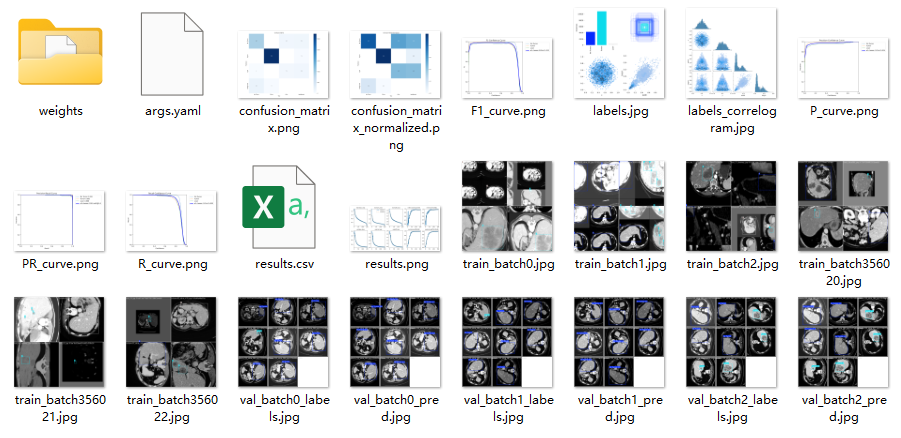
各损失函数作用说明:
定位损失box_loss:预测框与标定框之间的误差(GIoU),越小定位得越准;
分类损失cls_loss:计算锚框与对应的标定分类是否正确,越小分类得越准;
动态特征损失(dfl_loss):DFLLoss是一种用于回归预测框与目标框之间距离的损失函数。在计算损失时,目标框需要缩放到特征图尺度,即除以相应的stride,并与预测的边界框计算Ciou Loss,同时与预测的anchors中心点到各边的距离计算回归DFLLoss。这个过程是YOLOv8训练流程中的一部分,通过计算DFLLoss可以更准确地调整预测框的位置,提高目标检测的准确性。
训练结果如下:
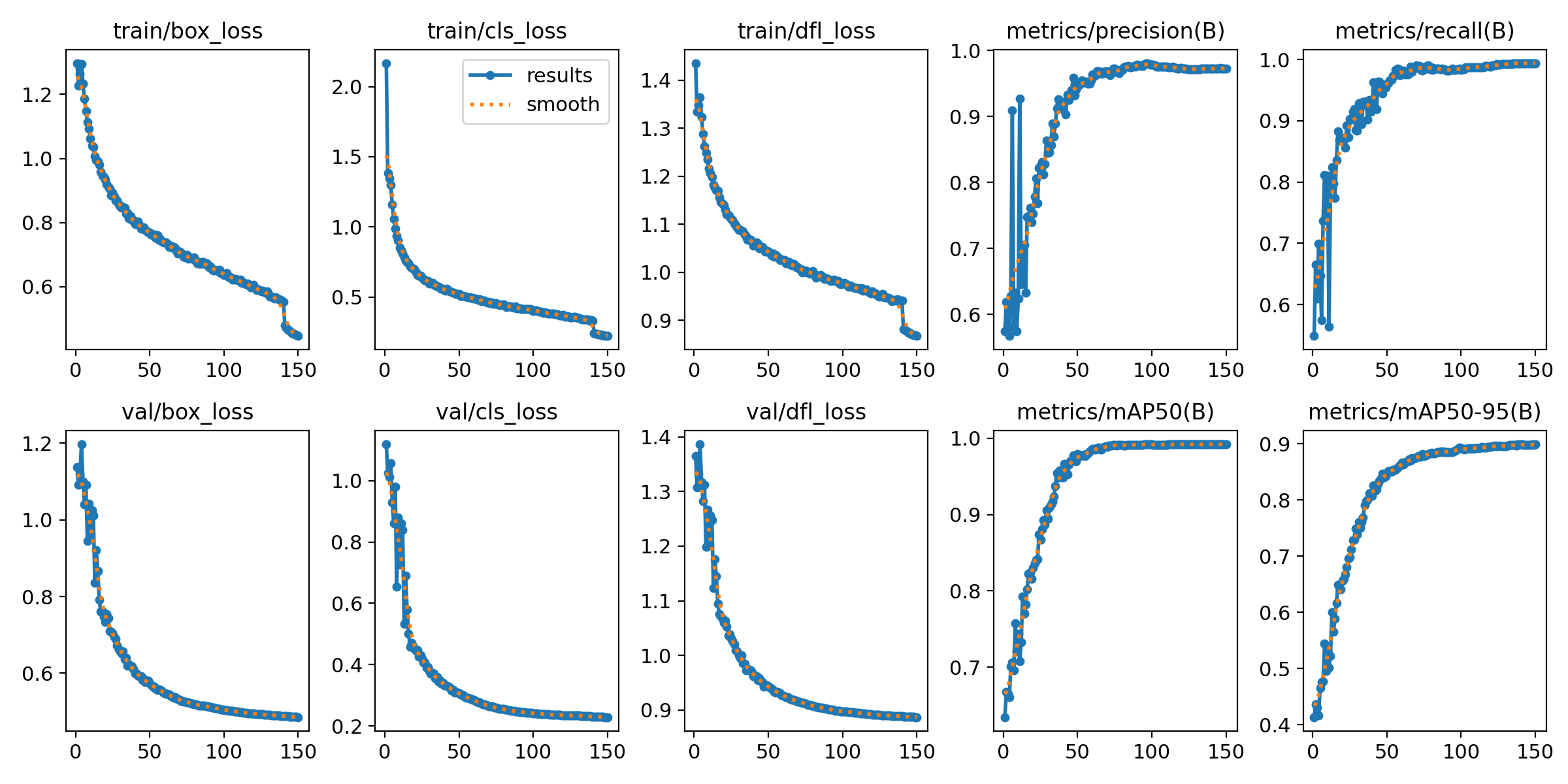
这张图展示了YOLOv8模型在训练和验证过程中的多个重要指标的变化趋势,具体如下:
train/box_loss:
(1)这是训练过程中边界框损失的变化。边界框损失用于衡量模型预测的目标框与实际目标框的差异。
(2)训练集上的边界框损失随着训练的进行逐渐下降。这表明模型在训练过程中对目标的定位精度不断提升。
train/cls_loss:
(1)这是训练集上的分类损失。分类损失衡量模型对目标类别的预测准确性。
(2)训练集上的分类损失逐渐下降,说明模型在训练过程中对类别的识别能力逐渐提高。
train/dfl_loss:
(1)这是分布聚焦损失(distribution focal loss),用于帮助模型对目标框的精确定位。
(2)训练集上的方向焦点损失(DFL)也呈下降趋势,这表明模型在边界框回归方面表现越来越好。
metrics/precision(B):
(1)这是训练集上的精度(precision)曲线。精度表示模型在检测到的目标中有多少是真正的目标。
(2)模型的精度随着训练轮次的增加逐渐提高,并趋于稳定,表明模型能够准确地预测目标类别。
metrics/recall(B):
(1)这是训练集上的召回率(recall)曲线。召回率表示模型检测出的真实目标的比例。
(2)召回率从初始训练时波动较大,逐渐稳定并接近1。这表明模型可以更好地检测出图像中的所有目标。
val/box_loss:
(1)这是验证集上的边界框损失曲线。
(2)验证集上的边界框损失也呈下降趋势,表明模型不仅在训练集上表现良好,在验证集上也能够准确定位目标。
val/cls_loss:
(1)这是验证集上的分类损失曲线。
(2)验证集上的分类损失也在逐渐降低,进一步证明模型的泛化能力较好。
val/dfl_loss:
(1)这是验证集上的分布聚焦损失曲线。
(2)验证集上的方向焦点损失逐渐下降,这和训练集的趋势一致,表明模型在回归任务上的表现持续提升。
metrics/mAP50(B):
(1)这是验证集上的mAP50曲线,表示在交并比阈值为0.5时模型的平均精度(mean Average Precision)。
(2)模型的mAP50(平均精度)随着训练轮次的增加迅速提升并保持在高水平,表明模型在50% IoU阈值下的检测精度表现良好。
metrics/mAP50-95(B):
(1)这是验证集上的mAP50-95曲线,表示在不同交并比阈值(从0.5到0.95)下模型的平均精度。
(2)mAP50-95逐渐提高,最后保持在约0.9,表示模型在更严格的IoU阈值下也能够维持较高的检测精度。
这组图图中的曲线显示了随着训练的进行,模型的损失逐步减少,精度和召回率稳步上升,mAP(平均精度)指标逐步提高并趋于稳定,说明模型的训练效果良好并具有较好的泛化能力。
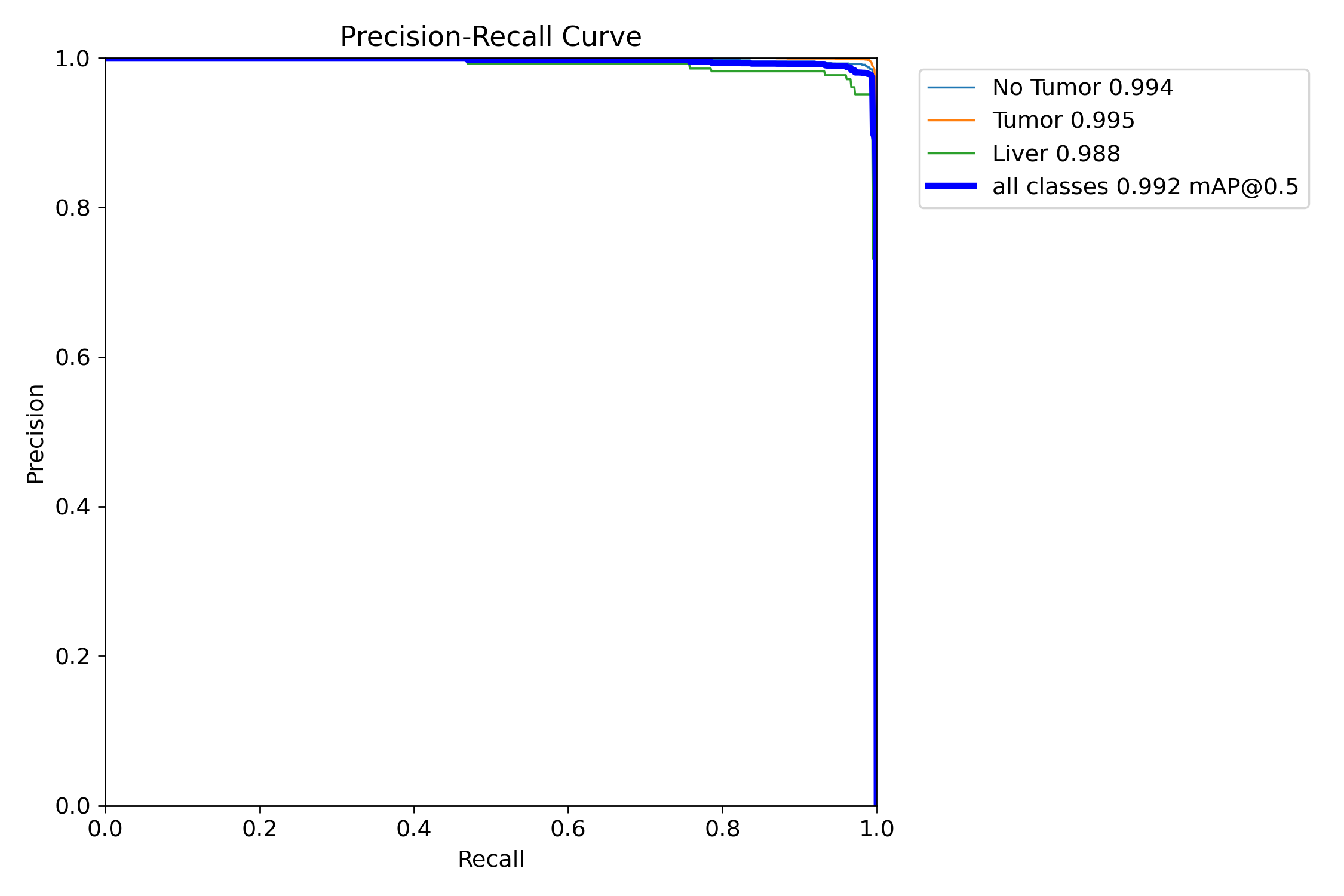
这张图展示的是 Precision-Recall 曲线,用于评估模型在不同类别下的检测性能。以下是详细解释:
各条曲线代表的含义:
不同颜色的曲线代表模型在各个类别上的精度和召回率之间的关系:
(1)蓝色曲线(No Tumor,0.994):表示对“无肿瘤”类别的精度-召回率曲线,mAP为0.994,表明该类别的检测性能非常高,模型几乎在所有召回率下都保持了很高的精度。
(2)橙色曲线(Tumor,0.995):表示对“肿瘤”类别的检测,mAP为0.995,说明模型对于“肿瘤”检测的精度和召回率表现非常好,几乎接近1。
(3)绿色曲线(Liver,0.988):表示对“肝脏”类别的检测,mAP为0.988,虽然略低于其他类别,但仍然非常接近1,表明模型对该类别的检测性能也很强。
(4)紫色曲线(all classes 0.992 mAP@0.5):表示所有类别整体的平均精度和召回率,mAP@0.5为0.992,这表明模型在所有类别上的总体表现非常出色。
总体表现:从曲线形状可以看出,所有类别的精度和召回率都接近完美,曲线在高精度和高召回率的范围内几乎达到顶端,这表明模型能够以较少的误报和漏报准确检测出不同类别的目标。
这张图显示了模型在不同类别下的高精度和高召回率,尤其是“肿瘤”和“无肿瘤”类别的检测性能极为优秀,说明该YOLOv8模型能够很好地识别肿瘤相关的目标。
4.检测结果识别
模型训练完成后,我们可以得到一个最佳的训练结果模型best.pt文件,在runs/train/weights目录下。我们可以使用该文件进行后续的推理检测。
imgTest.py 图片检测代码如下:
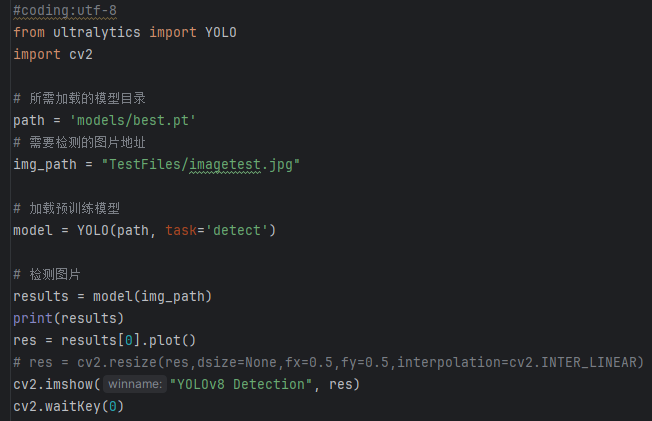
加载所需库:
(1)from ultralytics import YOLO:导入YOLO模型类,用于进行目标检测。
(2)import cv2:导入OpenCV库,用于图像处理和显示。
加载模型路径和图片路径:
(1)path = ‘models/best.pt’:指定预训练模型的路径,这个模型将用于目标检测任务。
(2)img_path = “TestFiles/0229.png”:指定需要进行检测的图片文件的路径。
加载预训练模型:
(1)model = YOLO(path, task=’detect’):使用指定路径加载YOLO模型,并指定检测任务为目标检测 (detect)。
(2)通过 conf 参数设置目标检测的置信度阈值,通过 iou 参数设置非极大值抑制(NMS)的交并比(IoU)阈值。
检测图片:
(1)results = model(img_path):对指定的图片执行目标检测,results 包含检测结果。
显示检测结果:
(1)res = results[0].plot():将检测到的结果绘制在图片上。
(2)cv2.imshow(“YOLOv8 Detection”, res):使用OpenCV显示检测后的图片,窗口标题为“YOLOv8 Detection”。
(3)cv2.waitKey(0):等待用户按键关闭显示窗口
此代码的功能是加载一个预训练的YOLOv8模型,对指定的图片进行目标检测,并将检测结果显示出来。
执行imgTest.py代码后,会将执行的结果直接标注在图片上,结果如下:
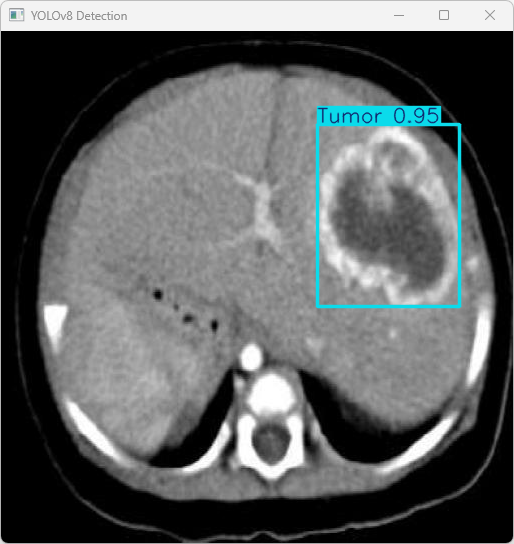
这段输出是基于YOLOv8模型对图片“imagetest.jpg”进行检测的结果,具体内容如下:
图像信息:
(1)处理的图像路径为:TestFiles/imagetest.jpg。
(2)图像尺寸为 512×512 像素。
检测结果:
(1)模型在该图片上检测到 1 个肿瘤(”1 Tumor”)
处理速度:
(1)预处理时间:4.8 毫秒
(2)推理时间:4.3 毫秒
(3)后处理时间:51.5 毫秒
模型在处理图片时非常高效,成功检测出 1 个肿瘤实例,并将结果保存到了指定目录。
运行效果
– 运行 MainProgram.py
1.主要功能:
(1)可用于实时检测目标图片中的肝脏肿瘤;
(2)支持图片、视频及摄像头进行检测,同时支持图片的批量检测;
(3)界面可实时显示目标位置、目标总数、置信度、用时等信息;
(4)支持图片或者视频的检测结果保存。
2.检测结果说明:
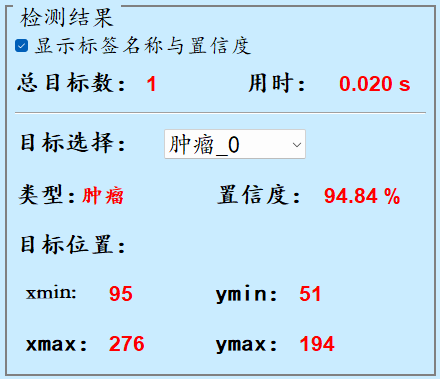
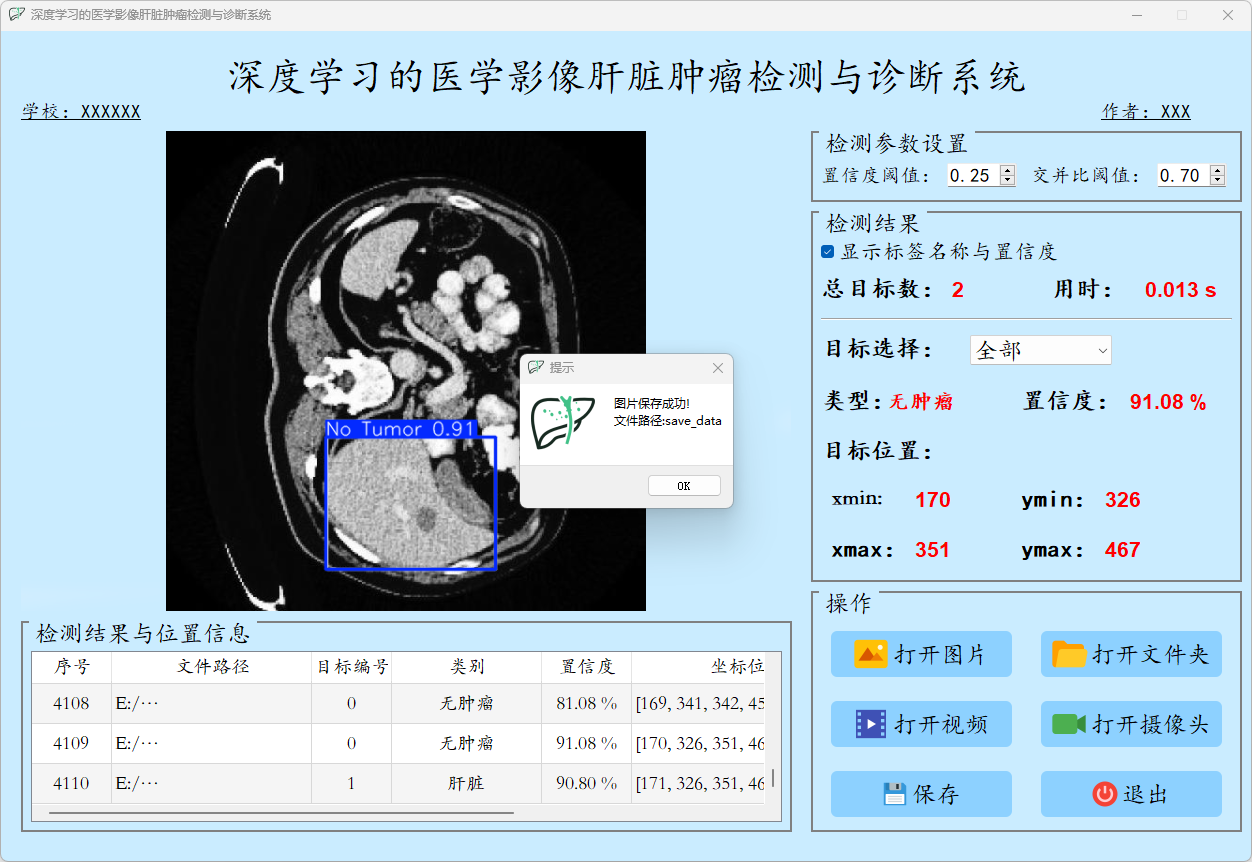
这张图表显示了基于YOLOv8模型的目标检测系统的检测结果界面。以下是各个字段的含义解释:
用时(Time taken):
(1)这表示模型完成检测所用的时间为0.020秒。
(2)这显示了模型的实时性,检测速度非常快。
目标数目(Number of objects detected):
(1)检测到的目标数目为1,表示这是当前检测到的第1个目标。
目标选择(下拉菜单):全部:
(1)这里有一个下拉菜单,用户可以选择要查看的目标类型。
(2)在当前情况下,选择的是“全部”,意味着显示所有检测到的目标信息。
类型(Type):
(1)当前选中的行为类型为 “肿瘤”,表示系统正在高亮显示检测到的“Tumor”。
置信度(Confidence):
(1)这表示模型对检测到的目标属于“肿瘤”类别的置信度为94.84%。
(2)置信度反映了模型的信心,置信度越高,模型对这个检测结果越有信心。
目标位置(Object location):
(1)xmin: 95, ymin: 51:目标的左上角的坐标(xmin, ymin),表示目标区域在图像中的位置。
(2)xmax: 276, ymax: 194:目标的右下角的坐标(xmax, ymax),表示目标区域的边界。
这些坐标表示在图像中的目标区域范围,框定了检测到的“肿瘤”的位置。
这张图展示了肝脏肿瘤的一次检测结果,包括检测时间、检测到的种类、各行为的置信度、目标的位置信息等。用户可以通过界面查看并分析检测结果,提升肝脏肿瘤诊断的效率。
3.图片检测说明
(1)肝脏
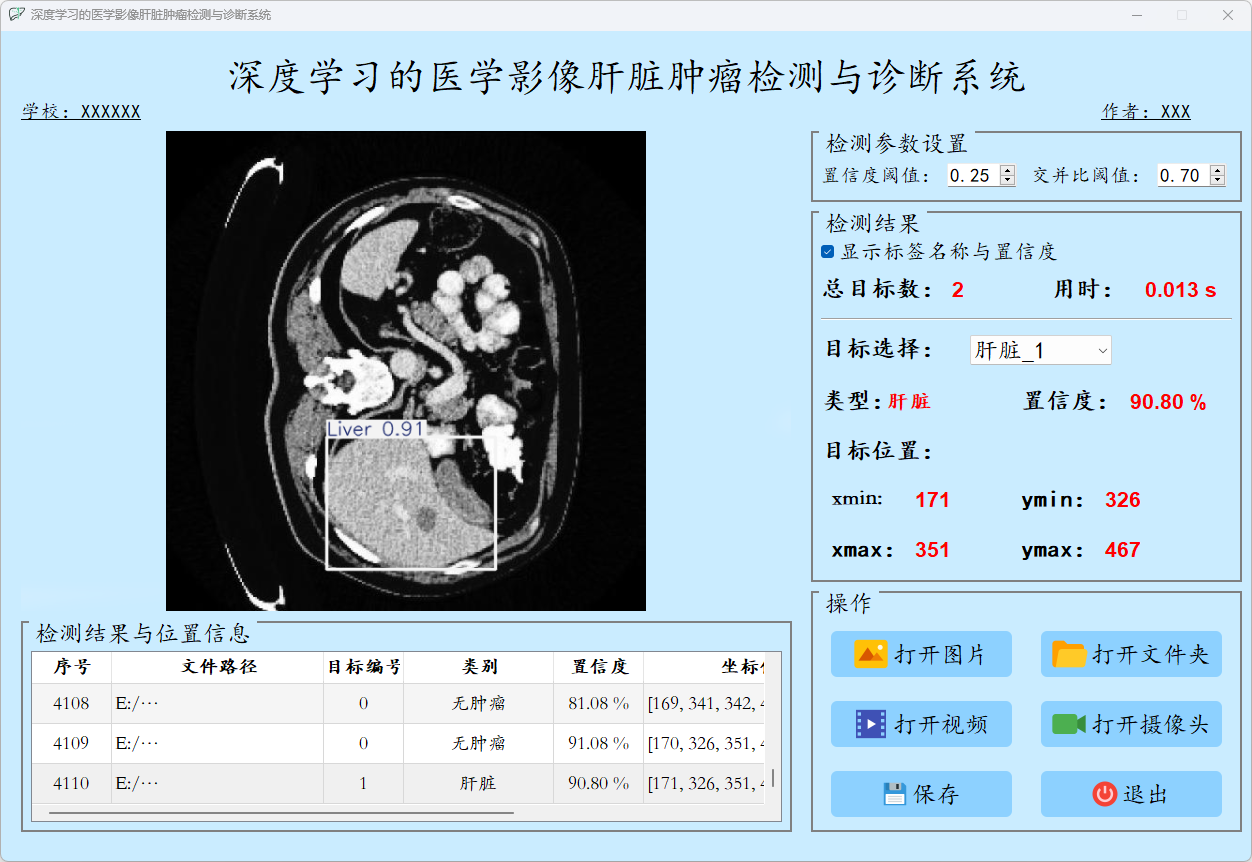
(2)肝脏无肿瘤
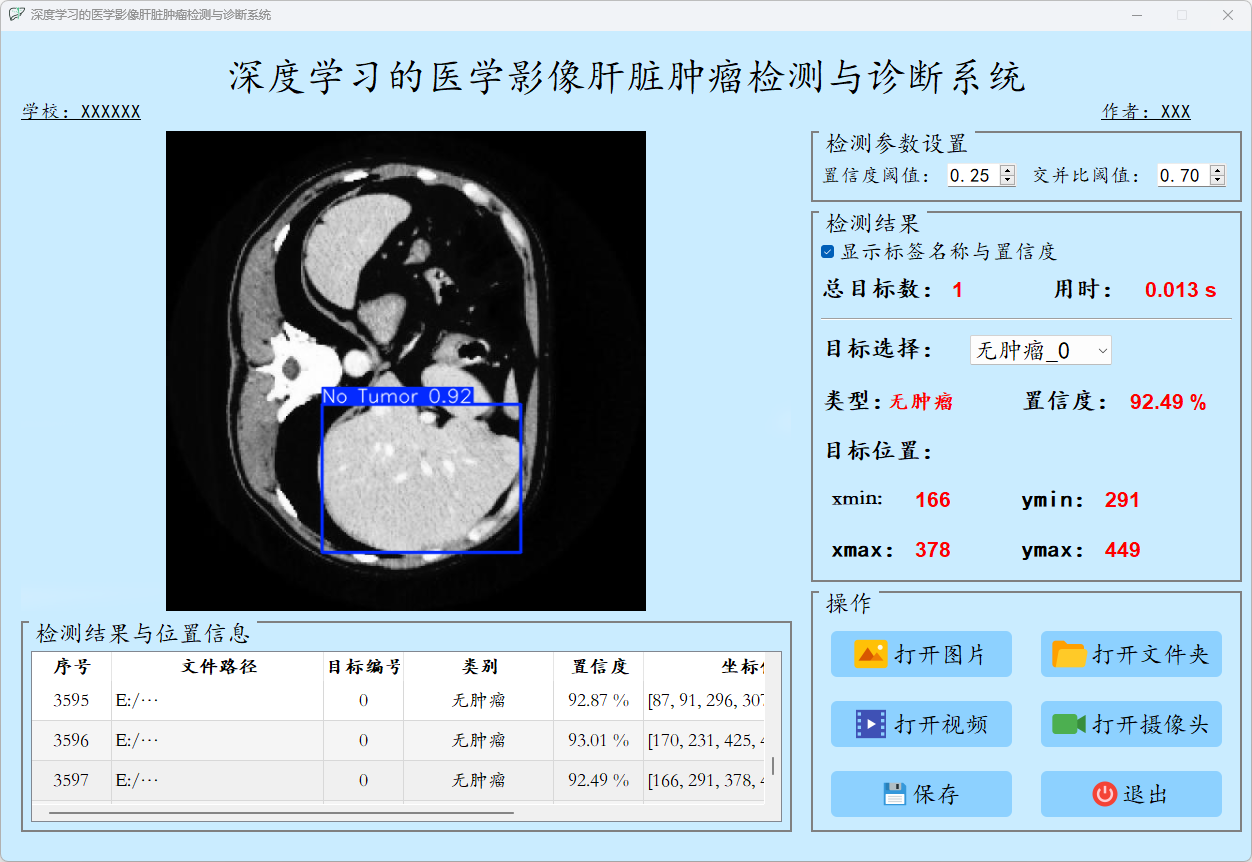
(3)肝脏肿瘤
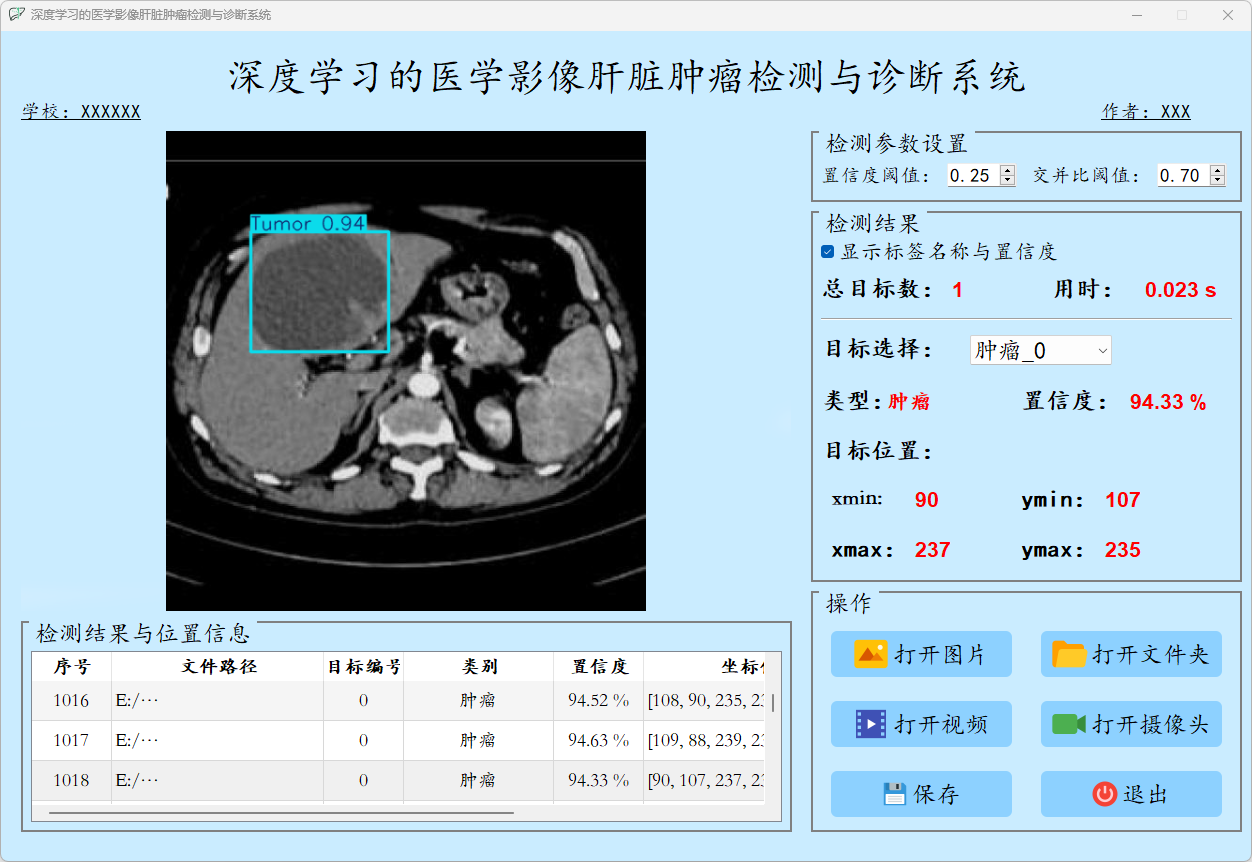
点击打开图片按钮,选择需要检测的图片,或者点击打开文件夹按钮,选择需要批量检测图片所在的文件夹。
操作演示如下:
(1)点击目标下拉框后,可以选定指定目标的结果信息进行显示。
(2)点击保存按钮,会对检测结果进行保存,存储路径为:save_data目录下。
检测结果:系统识别出图片中的肝脏病症,并显示检测结果,包括总目标数、用时、目标类型、置信度、以及目标的位置坐标信息。
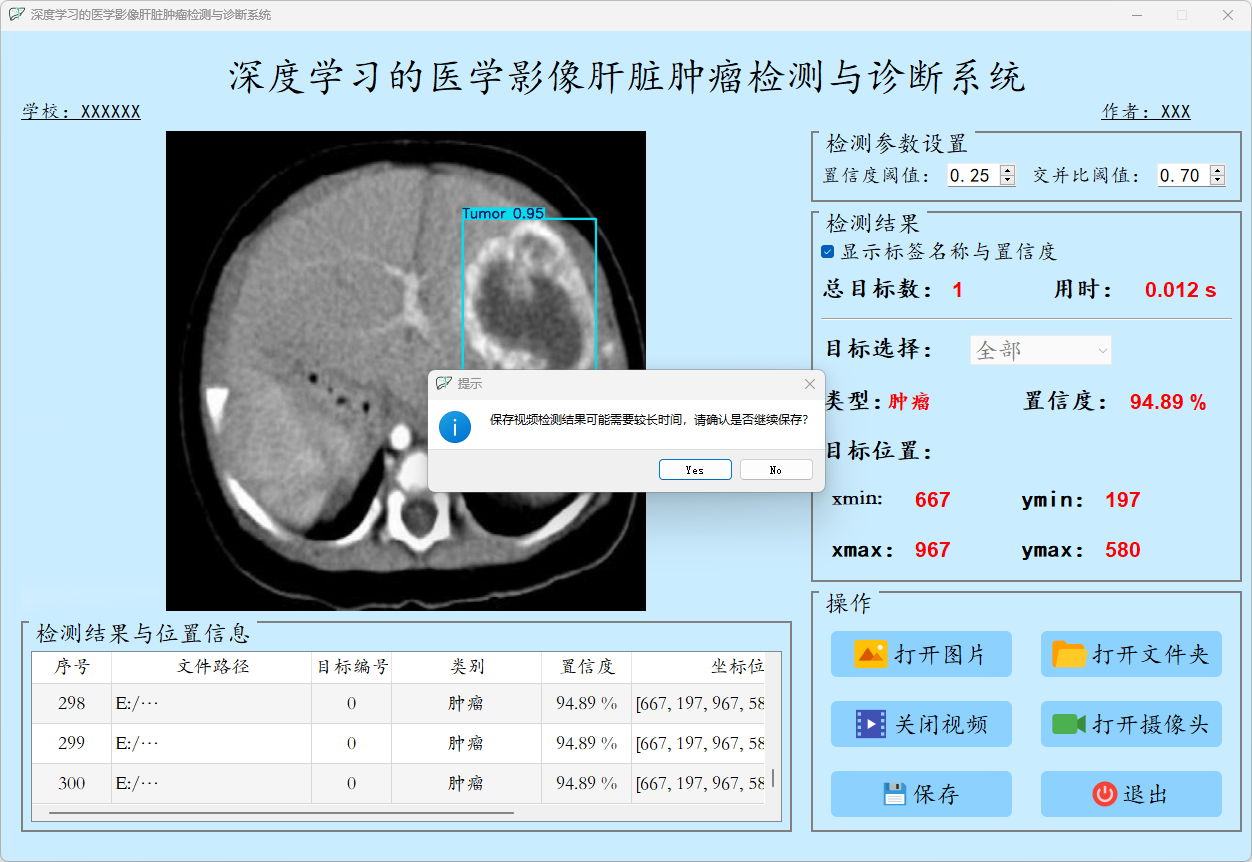
4.视频检测说明
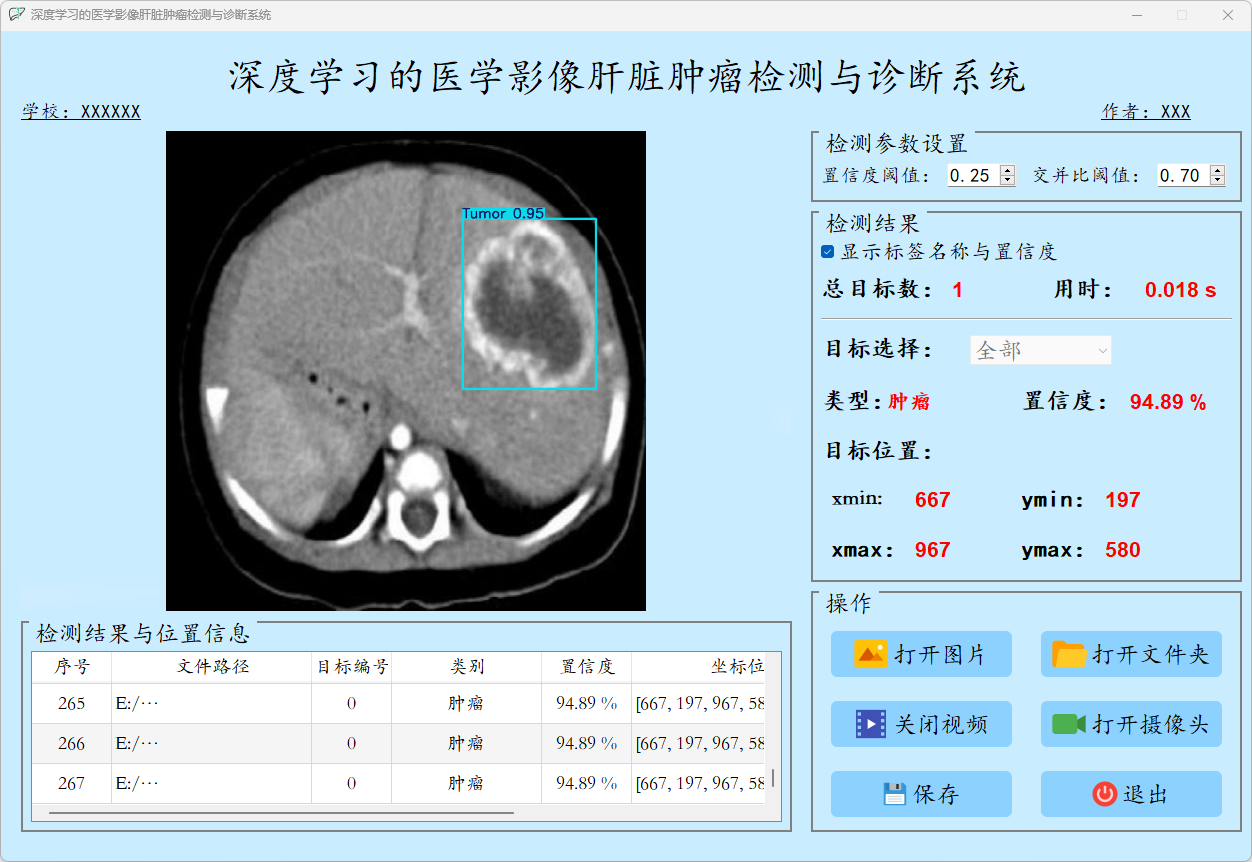
点击视频按钮,打开选择需要检测的视频,就会自动显示检测结果,再次点击可以关闭视频。
点击保存按钮,会对视频检测结果进行保存,存储路径为:save_data目录下。
检测结果:系统对视频进行实时分析,检测到肝脏病症并显示检测结果。表格显示了视频中多个检测结果的置信度和位置信息。
这个界面展示了系统对视频帧中的多目标检测能力,能够准确识别肝脏病症,并提供详细的检测结果和置信度评分。
5.摄像头检测说明
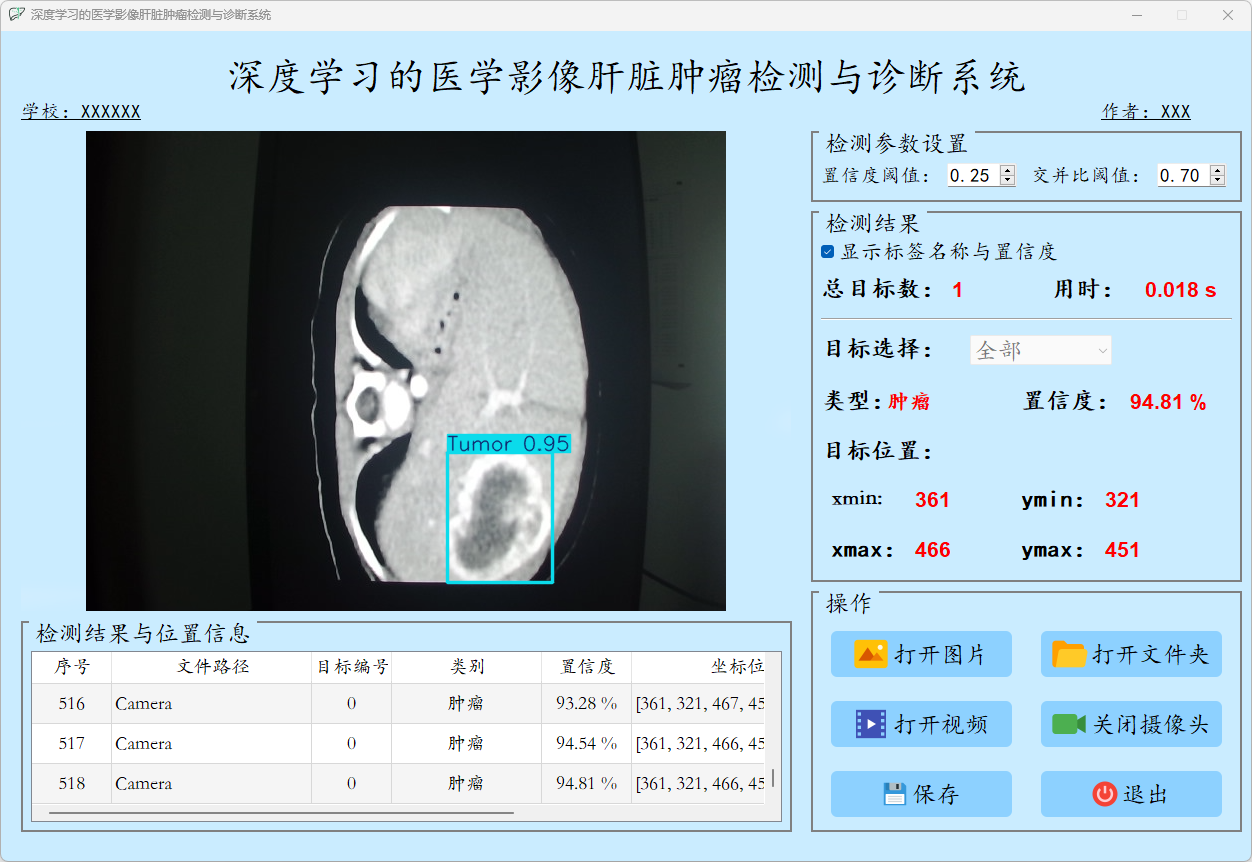
点击打开摄像头按钮,可以打开摄像头,可以实时进行检测,再次点击,可关闭摄像头。
检测结果:系统连接摄像头进行实时分析,检测到肝脏病症并显示检测结果。实时显示摄像头画面,并将检测到的行为位置标注在图像上,表格下方记录了每一帧中检测结果的详细信息。
6.保存图片与视频检测说明
点击保存按钮后,会将当前选择的图片(含批量图片)或者视频的检测结果进行保存。
检测的图片与视频结果会存储在save_data目录下。
保存的检测结果文件如下:
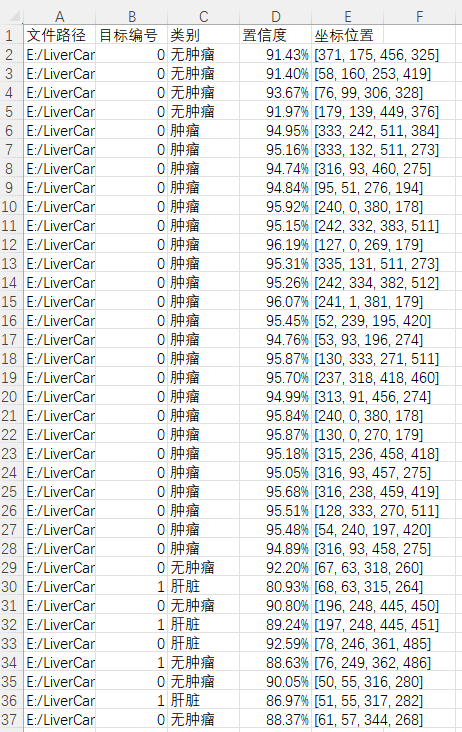
图片文件保存的csv文件内容如下,包括图片路径、目标在图片中的编号、目标类别、置信度、目标坐标位置。
注:其中坐标位置是代表检测框的左上角与右下角两个点的x、y坐标。
(1)图片保存
(2)视频保存
– 运行 train.py
1.训练参数设置

(1)data=data_yaml_path: 使用data.yaml中定义的数据集。
(2)epochs=150: 训练的轮数设置为150轮。
(3)batch=4: 每个批次的图像数量为4(批次大小)。
(4)name=’train_v8′: 训练结果将保存到以train_v8为名字的目录中。
(5)optimizer=’SGD’: 使用随机梯度下降法(SGD)作为优化器。
虽然在大多数深度学习任务中,GPU通常会提供更快的训练速度。
但在某些情况下,可能由于硬件限制或其他原因,用户需要在CPU上进行训练。
温馨提示:在CPU上训练深度学习模型通常会比在GPU上慢得多,尤其是像YOLOv8这样的计算密集型模型。除非特定需要,通常建议在GPU上进行训练以节省时间。
2.训练日志结果
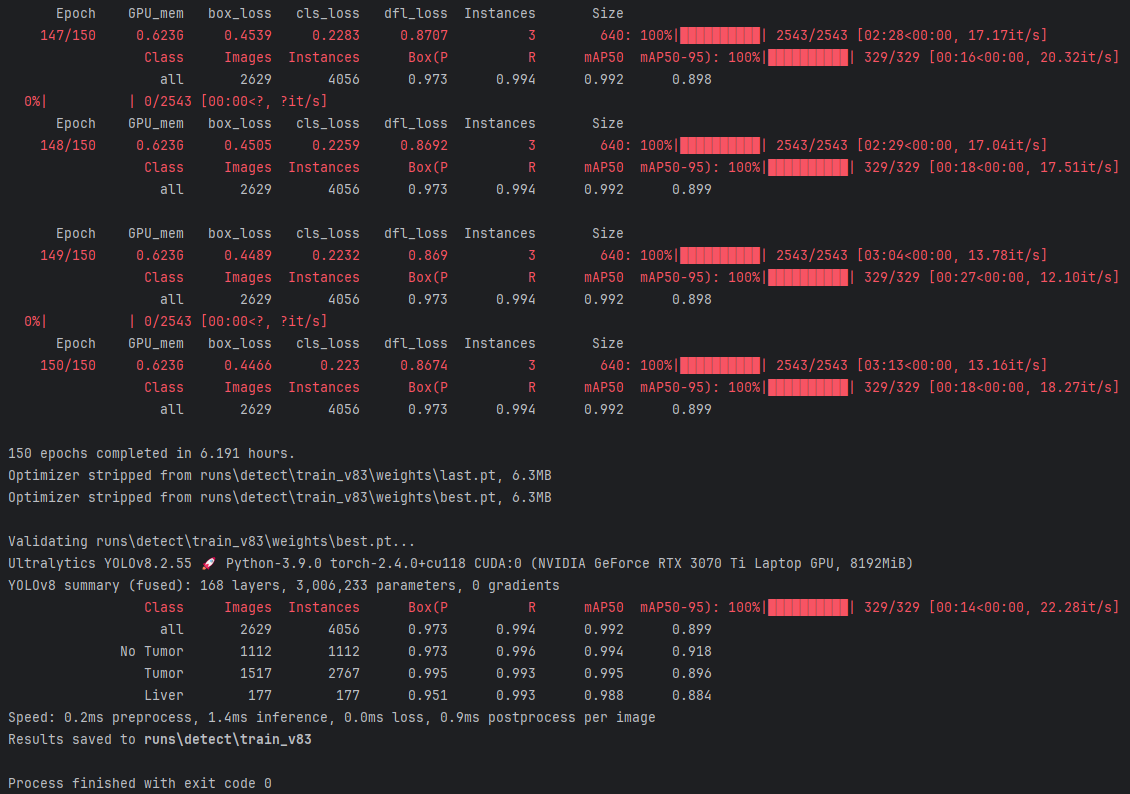
这张图展示了使用YOLOv8进行模型训练的详细过程和结果。
训练总时长:
(1)模型在训练了150轮后,总共耗时6.191小时。
模型验证:
1.使用验证集对模型进行评估,显示在不同类别(”No Tumor”、”Tumor”、”Liver”)下的mAP50和mAP50-95指标:
(1)No Tumor: mAP50-95为0.918。
(2)Tumor: mAP50-95为0.896。
(3)Liver: mAP50-95为0.884。
2.推理速度为0.2ms预处理、1.4ms推理、0.9ms后处理,整体表现出色。
结果保存:
(1)Results saved to runs\detect\train_v8:验证结果保存在 runs\detect\train_v8 目录下。
完成信息:
(1)Process finished with exit code 0:表示整个验证过程顺利完成,没有报错。
该图展示了YOLOv8模型在“无肿瘤”和“肿瘤”类别上表现非常好,尤其是在mAP50和mAP50-95指标上,精度和召回率都非常高,说明其检测效果和泛化能力都很强。